Gradiente Proyectado Descafeinado
Mariano Rivera
octubre 2018
Programas cuadráticos con restricciónes de cota
Los programas cuadráticos con restricciónes de cota (Bounded Quadratic Programming, B-QP) son problemas de optimización con restricciones [1]. Éstos son populares en problemas en tareas relacionadas con reconocimietno de patrones, aprendizaje de máquina, procesamiento de imágenes y visión por computadora. Su forma general es
(1)
donde es una matriz Hessiana (simétrica), y son vectores de iguales dimensiones que y definen la cota inferior y superior de los valores válidos para . El conjunto de puntos válidos definen la región factible .
[1] De Angelis P.L., Toraldo G. (2008) Quadratic Programming with Bound Constraints. In: Floudas C., Pardalos P. (eds) Encyclopedia of Optimization. Springer, Boston, MA
Ejemplos de programas cuadráticos con restricciónes de cota
Mínimos Cuadrados no Negativos (NNLS)
(2)
En este caso tenemos que
(3)
por lo que (2) equivale a la forma (1) con , , y .
Casos de problemas de NNLS son los problemas de Persecusión de Bases (Basis Pursuit). En estos problemas se cuenta con un diccionario de funciones y se busca encontrar la combinación lineal (suma pesada-positiva) de dichas funciónes para ajustar una señal . Generelmente el diccionario es sobredeterminado (tenemos mas funciones componentes que variables en : ) por lo que el prolema debe ser regularizado:
(4)
donde las columnas de A son las funciones , (), es un parámetro de penalización (regularización Ridge) que promueve que usar pocas funciones para en solución.
Para mas sobre regularización de paramétros por contracción (shrinkage), ver Regularización Ridge, LASSO y ElasticNet.
[2] D. Donoho, I. Johnstone, J. Hoch, and A. Stern. Maximum entropy and
the nearly black object. Journal of the Royal Statistical Society Series B,
54:41–81, 1992.
[3] D. Kim, S. Sra, and I. Dhillon. Tackling box-constrained convex opti- mization via a new projected quasi-Newton approach. SIAM Journal on Scientific Computing, 32:3548–3563, 2010.
Factorización no negativa de matrices (NNMF)
Sea una matrix de rango con todos sus componentes no-negativos: . Entonces puede factorizarse como el producto de dos matrices nonegativas [4]:
(5)
con y . La factorización se obtienen resolviendo
(2)
Note que este problema es no cuadrático debido al producto entre las incógnitas. Si no se conoce o se predende calcular una factorización mas comprimida, entonces se propone una valor para mediante la solución de (2) obtenemos una buena aproximación.
Importantes propiedades de NNMF han sido reportadas en [5,6]
[5] C. Ding et al. On the equialence of nonnegative matrix factorization and spectral clustering, Proc. SIAM Int. Conf. Data Mining, 2005
[6] M. Slawski and M. Matthias. Non-negative least squares for high-dimensional linear models: Consistency and sparse recovery without regularization. Electron. J. Statist. 7 (2013), 3004–3056. doi:10.1214/13-EJS868.
Máquinas de Vectores de Soporte (SVM)
Sea un vector que corresponde al -ésimo dato tal que la -ésima entrada corresponde a la -ésima característica. Luego arreglamos los datos de la forma
de tal que cada dato corresponde a un renglón de la matrix y tenemos una etiqueta asociada a cada dato. LA etiqueta indica la clase a que pertenece. Luego denotamos el vector de etiquetas por
La SVMs para clasificación binaria se formulan como un QP con resticciones de cota y una restricción de igualdad. El problema a resolver esta dado por
(6)
donde es una matrix semidefinida positiva con elementos dados por
(7)
para el caso de clases linelamente separables y
(8)
para el caso de clases no-linelamente separables, donde es una función Kernel qel mapeo de datos 's a un espacio de mayor dimensión: ; espacio donde se espera que las clases sean linelamente separables. Finalmente permite hacer la clasificación robusta a outliers. En el caso en que las calses sean estrictamente linealmente separables ; es decir no hay restricción de cota superior.
Para más sobre SVMs ver la siguiente liga, el artículo original [7], el tutorial [8].
[7] Cortes, C. & Vapnik, V. (1995). Support-vector network. Machine Learning, 20, 1–25.
[8] M.A. Hearst et a., Support vector machines, IEEE Int. Syst., 13(4), pp. 18-28 , 1998, DOI: 10.1109/5254.708428.
El Gradiente Protectado como Motivación
[5] J. Nocedal and S. Wright, Numerical Optimization, 2nd Ed. Springer, 2006.
El algoritmo de gradiente proyectado puede entenderse a partir de la siguiente figura.

En el panel (a) se asume que iniciamos en un punto actual dentro de la región factible (región rectangular en verde). Si usamos una estrategia de máximo descenso, sin considerar las restricciones, entonces el paso estaría definido por Sin embargo el nuevo punto no sería factile. Para evitar ello, el paso (gradiente con tamaño de paso) es proyectado a la región factible usando
El paso proyectado forma un camino recto a pedazos como lo muestra el Panel (b). Sobre el camino proyectado se busca el primer mínimo mediante una inspeción de tramo por tramo. El primer mínimo se muestra en el Panel © que es el lugar en que el camino proyectado es tangente a una curva (o superficie) de nivel.
El algoritmo de Gradiente Proyectado tienen un paso más que mejora su convergencia. Una vez que se determina el primer mínimo en lo largo del camino proyectado, se fijan las variables que han activado restricciones (denominadas variables activas) y se procede a minimizar, aproximadamente, en el sub-espacio definido por las variables no activas.
Decaf Gradiente Protectado
Cuando nos referimos a la versión descafeinada de un algoritmo nos referimos a una versión más fácil de implementar y, si bien es tan eficiente en términos de convergencia que la versión original, si grantiza convergencia a la solución. Una versión DECAF puede ser muy útil para desarrollar prototipos; en la etapa en que aun estamos haciendo pruebas a nuestros modelos.
El Gradiente Proyectado Descafeinado se ilustra en la siguiente figura.

La estrategia de gradiente proyectado re resume en iterar:
-
Partimos de un punto factible , y calculamos la predicción de actualización sin considerar las restricciónes,
-
Proyectamos esa actualización a la región factible, esto se conigue recortando (clipping) las variables que salen de la región factible.
-
Aplicamos unos descenso de gradiente en las variables que no fueron “recortadas”.
Mas formalmente lo implementamos como sigue:
Sea un vector con unos en todas sus entradas y de las mismas dimensiones que , luego
Iterar los siguientes pasos hasta convergencia
-
Calcular el gradiente de la función objetivo
-
Calcular la dirección de descenso:
-
Reinicializar cada iteraciones:
if t%k==0: m=1 -
Actualizar los parámetros
donde es el producto elemento a elemento (producto de Hadamard: ). -
Detectar los índices de las entradas que violan la restricción:
-
Recortar (Clipping) las variables que se salen de la región factible: hacer
-
Recortar : hacer
Ejemplo 1: Aproximación mediante NN-RBF
Dada la función suave
donde es una constante que hace que y para ; con
Resolver
donde
es un diccionario de funciones base
y es el -ésimo átomo (función base), que es un vector columna y se define por
import numpy as np
import matplotlib.pyplot as plt
x = np.array(range(100))
def f(x):
y=np.sin(x/7)+2.*np.cos((x-70)/20)
return y-np.min(y)
def gss(x, media, s):
return np.exp(-(x-media)**2/(2.*s**2))
def calculaPhi(x, numRBF=10, sigma=6):
ndata=len(x)
paso = np.ceil(ndata/numRBF)
medias = np.arange(0,ndata+paso,paso)
Phi=[]
for media in medias:
phi= gss(x,media,sigma)
Phi.append(phi)
return np.array(Phi).T
def calculaGradf(y, Phi, theta):
grad = Phi.T@(Phi@theta-y)
return grad
# señal
y=f(x)
# Diccionario
Phi=calculaPhi(x=x, numRBF=12, sigma=6)
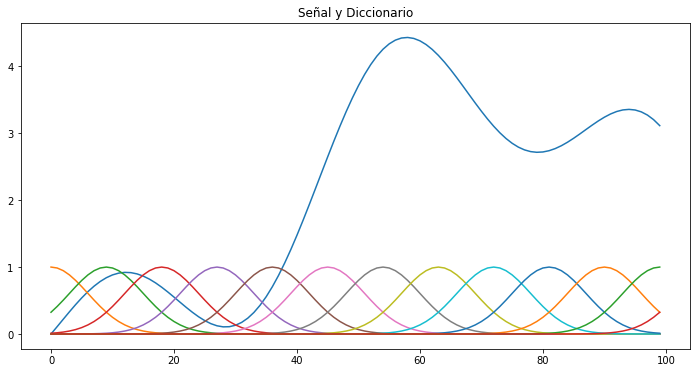
Deiccionario y función a aproximar
plt.figure(figsize=(12,6))
plt.plot(y)
n,m= Phi.shape
for i in range(m):
plt.plot(Phi[:,i])
plt.title('Señal y Diccionario')
plt.show()
Desceso de gradiente simple sin restricciones
N=100
theta = np.ones(m)
alpha=0.01
for i in range(N):
grad = calculaGradf(y, Phi, theta)
theta=theta-alpha*grad
print('grad=\n', grad)
print('theta=\n', theta)
plt.figure(figsize=(12,6))
plt.plot(y, 'r')
for i in range(m):
plt.plot(theta[i]*Phi[:,i])
haty= Phi@theta
plt.plot(haty, 'go')
plt.title('Approximación RBF')
plt.show()
grad=
[ 0.15576335 -0.13285693 0.06640374 -0.00863362 -0.01732008 0.01881689
-0.01368835 0.0102288 -0.00404902 -0.01093976 0.03037573 -0.03781717
-0.00632543]
theta=
[-0.07728272 0.6386445 0.57739821 -0.17022554 0.1908662 1.63681606
2.84901629 2.64892047 1.72460445 1.43476044 2.09451563 2.04728421
1.17367164]
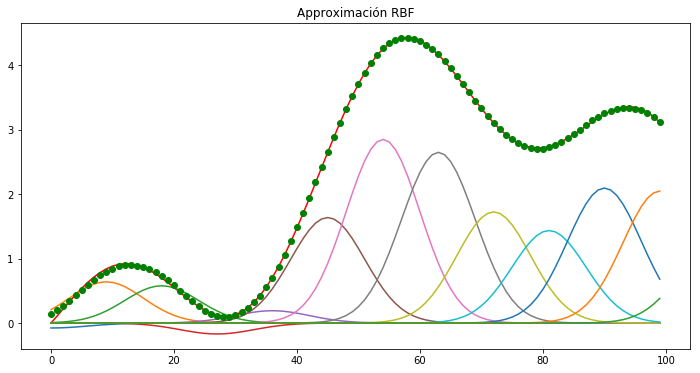
Como podemos observar, la aproximación (puntos verdes) se ajusta muy bien a la función original (línea roja)
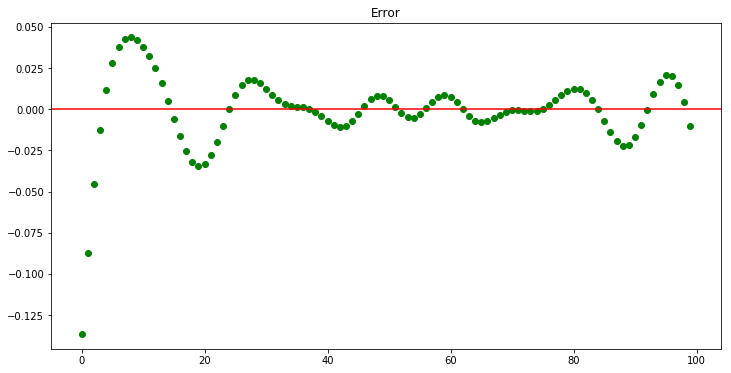
En la siguiente figura desplegamos el error en cada punto.
plt.figure(figsize=(12,6))
plt.plot(y-haty, 'go')
plt.axhline(y=0, color='r', linestyle='-')
plt.title('Error')
plt.show()
Descenso de gradiente proyectado descafeinado
Ahora la implementación del GP-Decaf para NN-RBF
N=300
theta = np.ones(m)
alpha=0.01
msk = np.ones(theta.shape)
K=10
for i in range(N):
grad = msk*calculaGradf(y, Phi, theta)
theta=theta-alpha*grad
# proyeccion
msk[theta<0]=0
theta[theta<0]=0
if i%K==0:
msk = np.ones(theta.shape)
print('norma del grad=\n', grad@grad)
print('theta=\n', theta)
plt.figure(figsize=(12,6))
plt.plot(y, 'r')
for i in range(m):
plt.plot(theta[i]*Phi[:,i])
haty= Phi@theta
plt.plot(haty, 'g*')
plt.title('')
plt.show()
norma del grad=
0.000127934053097
theta=
[ 0. 0.65809328 0.46698228 0. 0.0736745 1.70112498
2.81665557 2.66632238 1.71049321 1.45282892 2.07062272 2.07227976
1.16924487]
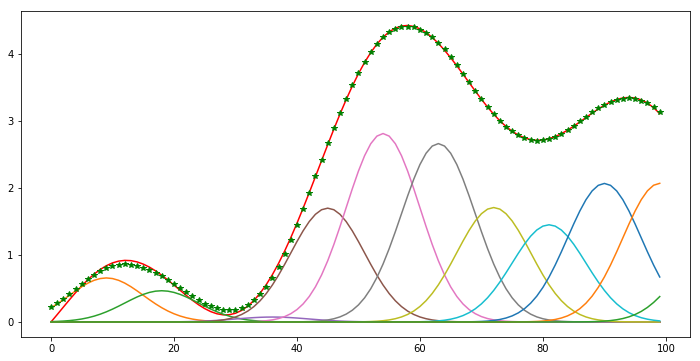
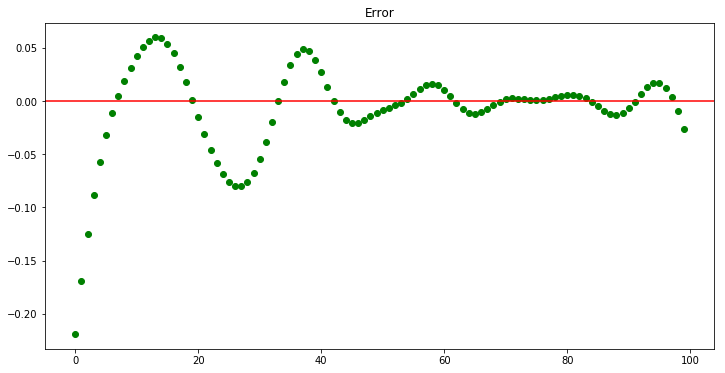
Note que no hay valores negativos en los coeficientes calculados, de hecho la solución no negativa requiere de 2 (ver los zeros) funciones base menos que la no retringida. Los errores se concentran al inicio donde la estimación esta por arriba de los datos y no es posible reducir la aproximación sin usar coeficientes negativos.
plt.figure(figsize=(12,6))
plt.plot(y-haty, 'go')
plt.axhline(y=0, color='r', linestyle='-')
plt.title('Error')
plt.show()
Ejemplo 2: SVMs
En este ejercicio entrenaremos una máquina de vectores de soporte para clasificación binaria de las dos primeras clases () de la base de datos IRIS.
La base de datos IRIS esta diponible en el paquete de python scikit-learn y consta de 4 clases, de las cuales seleccionamos las dos primeras, cada datos consta de 4 rasgos medidos a flores iris:
- Largo del sépalo
- Ancho del sépalo
- Largo del pétalo
- Ancho del pétalo
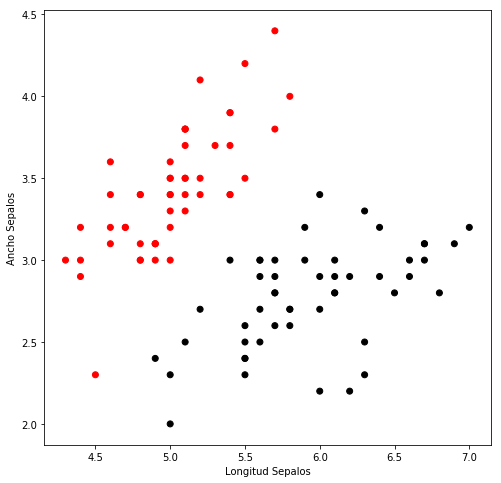
Se seleccionaron esas clases y rasgos por ser en el linealmente separables como se muestra en los puntos gráficados.
La estrategia utilizada es optmizar el Lagrangiano Aumentado [5] con la restricción de igualdad y con Decaf Gradiente Proyectado se implementa la restricción de cota (no-negatividad).
Lectura de los Datos
%matplotlib inline
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn import datasets
iris = datasets.load_iris()
print("llaves :", iris.keys())
print('número de muestras :', iris['data'].shape[0])
print('número de caractéristicas : ', iris['data'].shape[1])
llaves : dict_keys(['data', 'target', 'target_names', 'DESCR', 'feature_names'])
número de muestras : 150
número de caractéristicas : 4
idx= iris.target<2 # primeras dos clases
y= iris.target[idx]
y =2*y -1 # {-1, 1}
#-[1] # cambiamos a un dato su etiqueta
x = iris.data[idx, :2] # primeras dos características
plt.figure(figsize=(8, 8))
plt.scatter(x[:, 0],
x[:, 1],
c=Y,
cmap=plt.cm.flag)
plt.xlabel('Longitud Sepalos')
plt.ylabel('Ancho Sepalos')
plt.show()
Lagrangiano y su gradiente
def LagrangianAug(Q, y, theta, Lambda, rho):
return 0.5* theta.T@Q@theta - np.sum(theta) - Lambda*constricc + 0.5*rho* constricc**2
def gradLagrangianAug(Q, y, theta, Lambda, rho):
return Q@theta - 1.0 - (Lambda - rho*y.T@theta)*y
theta = np.ones(y.shape)
nIter=150000
alpha=0.001
if y.shape !=(100,1):
y= np.expand_dims(y, axis=1)
Lambda = 1.0
rho = 1/1000.
theta = np.ones((x.shape[0],1))
msk = np.ones(theta.shape)
K=5
Q = (y*x)@(y*x).T
for i in range(nIter):
grad = msk*gradLagrangianAug(Q=Q, y=y, theta=theta, Lambda=Lambda, rho=rho)
theta=theta-alpha*grad
# proyeccion
msk[theta<0]=0
theta[theta<0]=0
if i%K==0:
msk = np.ones(theta.shape)
if rho<100:
rho*=1.01
Lambda=Lambda-rho*y.T@theta
print('norma del grad=\n', grad.T@grad)
norma del grad=
[[ 0.00838404]]
Valor de la restricción de igualdad
np.sum(y*theta)
-6.7296752614964817e-09
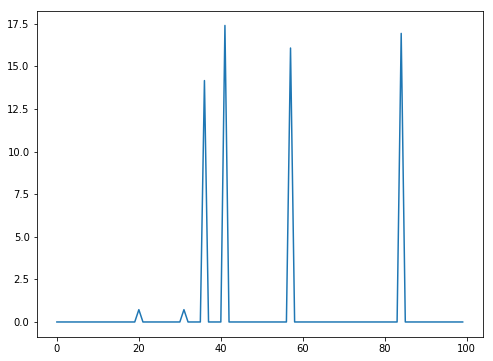
Valor de los multiplicadores de Lagrange que indican los vectores soporte detectados
plt.figure(figsize=(8,6))
plt.plot(theta)
plt.show
<function matplotlib.pyplot.show>
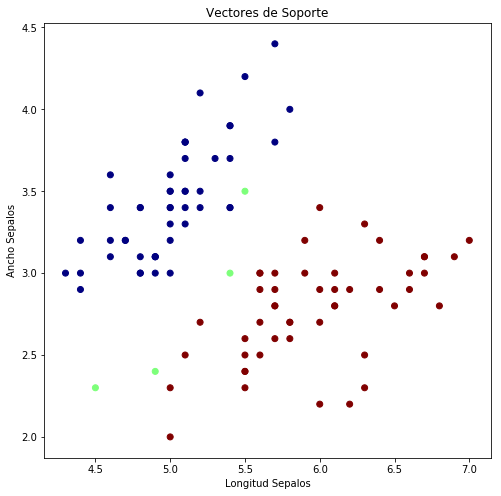
idx = theta>5.0
y[idx]=0
plt.figure(figsize=(8, 8))
plt.scatter(x[:, 0],
x[:, 1],
c=(y[:,0]+1.0)/2.0,
cmap=plt.cm.jet)
plt.xlabel('Longitud Sepalos')
plt.ylabel('Ancho Sepalos')
plt.title('Vectores de Soporte')
plt.show()
Ejercicio 1. Implementar NNMF usando decaf-pg
Ejercicio 2. Implementar la versión decaf-nesterov para el problema de SVM