Análsis de Componentes Principales
(Principal Component Analysis, PCA)
Mariano Rivera
Mayo 2017
-
PCA es un un método de transformación lineal de los datos que decorrelacional variables Gaussianas multivariadas
-
Inventado por el matemático-bioestadístico Pearson [1].
-
Es un método exploratorio que se a popularizado para reducir la dimensión de variables multivaluadas
[1 ] Pearson, K. (1901). “On Lines and Planes of Closest Fit to Systems of Points in Space,” Philosophical Magazine. 2 (11): 559–572. doi:10.1080/14786440109462720.
%matplotlib inline
#%matplotlib notebook
import numpy as np
import sklearn as skl
import matplotlib.pyplot as plt
from PIL import Image
#---------------------------------------------------------------------
def printArray(X, precision=3, suppress = True):
printOptionsOld = np.get_printoptions()
np.set_printoptions(precision=precision, suppress=suppress)
print(X)
np.set_printoptions(**printOptionsOld)
#---------------------------------------------------------------------
Datos Sintéticos
Para introducir el tema, generamos muestras con distribuciones Gaussianas multivariadas; i.e., cada dato en un vector.
Distribución Gaussiana multivariada decorrelacionada
El primer caso, corresponde a una Gaussiana multivariada no correlacionada con dimensión . Sea el -ésimo dato de la muestra con . Entonces,
(1)
donde y son la media y la variaza de la variable -ésima, respectivamente.
Luego la muestra esta dada por:
(2)
con
La covarianza entre las variables se obtienen mediante
(3)
donde son las medias muestrales (estimación de ).
En forma matricial este cálculo se obtienen
(4)
con
Operación simbólica en Python usando la librería Sympy
from sympy import *
init_printing()
# x'sa minúsculas son las entradas de la matriz de datos
# Xi mayúsculas es cada observacion
# Xses ma muestra toital (todos los datos ordenados por renglon), ecuación (2)
X1 = Matrix(symbols('x1(1:4)'))
X2 = Matrix(symbols('x2(1:4)'))
X3 = Matrix(symbols('x3(1:4)'))
X4 = Matrix(symbols('x4(1:4)'))
Xs = Matrix([X1.T, X2.T, X3.T, X4.T])
Xs
#matriz de covarianzas (escalada por m), ecuación (4)
Xs.T*Xs
Que es lo mismo que
# simbólicamente
(X1*X1.T+X2*X2.T+X3*X3.T+X4*X4.T)
Forma estandarizada (normalizada) de una variable aleatoria
Esta forma se obtienen dividiendo cada variable entre la respectiva varianza:
(5)
donde son las desviaciones estándares muestrales (estimación de ).
Y la matriz equivalente a (3) se denomina Matriz de Correlacion:
(6)
# Gaussiana tri-variada no-correlacionada
m = 10000 # número de datos (tamaño de la mustra)
n = 3 # dimensión de cada dato
Med = np.array([10, 3, -.5]) # vector de medias
Cov = np.array([[5, 0, 0], [0, 0.1, 0],[0, 0, 3]]) # Covarianza diagonal
X = np.random.multivariate_normal(Med, Cov, m)
from mpl_toolkits.mplot3d import Axes3D
def plotData3D(X, projection='3d'):
fig = plt.figure(figsize=(12,4))
ax = fig.add_subplot(131, projection=projection)
ax.scatter(X[:,0],X[:,0],X[:,2])
ax.view_init(elev=10,azim=-30) # Set rotation angle to 30 degrees
ax.set_xlabel('$x_1$')
ax.set_ylabel('$x_2$')
ax.set_zlabel('$x_3$')
ax.set_title('originales')
X -= np.mean(X, axis=0) # resta la media
ax = fig.add_subplot(132, projection=projection)
ax.scatter(X[:,0],X[:,0],X[:,2])
ax.view_init(elev=10,azim=-30) # Set rotation angle to 30 degrees
ax.set_xlabel('$x_1$')
ax.set_ylabel('$x_2$')
ax.set_zlabel('$x_3$')
ax.set_title('centrados')
X /= np.std(X, axis=0) # divide entre la varianza
ax = fig.add_subplot(133, projection=projection)
ax.scatter(X[:,0],X[:,0],X[:,2])
ax.view_init(elev=10,azim=-30) # Set rotation angle to 30 degrees
ax.set_xlabel('$x_1$')
ax.set_ylabel('$x_2$')
ax.set_zlabel('$x_3$')
ax.set_title('normalizados')
plt.show()
plotData3D(X)
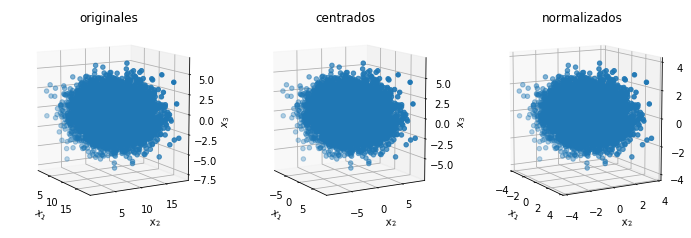
Aunque las tres figuras se ven iguales, nótese que los datos de originales no estan centrados (no tienen media cero) y solo solo normalizados tienen igual rango dinámico en las tres componentes de las variables
Esto la diferencia entre de los eigenvalores calculados usando datos centrados vs normalizados se muestra en los códigos que siguen, Notese que los eigenvectores prácticamente no son afectados, mayor aun para estos datos no correlacionados. Compárese con los datos correlacionados
Descomposición Valores Propios (Eig)
Sea la matriz de covarianza, definida semipositiva y simética, entonces, su descomposición eigenvalores propios resulta en la forma:
(7)
donde es una matriz diagonal con entradas con los eigenvalores ordenados descendentemente: ; las columnas de la matriz ortonormal
son los vectores propios tal que el eigenvector esta asociado al eigenvalor . Más aun, el eigenvalor corresponde a la varianza de los datos a los largo del eje definido por el eigenvector .
En caso de variables Gaussianas no correlacionadas, los eigenvectores coincidiran con los la base canónica; i.e.
Note que, dada la forma de la descomposición (7), la matriz a descomponer tendrá que ser necesariamente cuadrada. Algunas matrices cuadradas implicarán factorizaciones complejas (en el sentido de reales e imaginarios). Pero todas la matrices reales, semipositivas definidas y simétricas (como las de covarianza) se pueden descomponer, que son las que nos interesan.
Descomposición Valores (SVD)
Sea una matriz de datos, no necesariamente cuadrada () por lo que será en general no-definida y no-simética.
Entonces, su descomposición en eigenvalores no será posible. Pero, existe una forma alterna de descomposición, la descomposición en valores singulares :
(8)
donde:
- Los vectores singulares izquierdos de , , son los eigenvectores de :
(9)
- Los vectores singulares derechos de , , son los eigenvectores :
(10)
- una matriz diagonal con entradas con los valores singulares ordenados descendentemente: ; Sus valores corresponden al cuadrado de los eigenvalores no-zero de ambas, y .
# Code source: Mariano Rivera
# mayo 2017
#-------------------------------------------------------------------
def EigSvd(X):
X = X - np.mean(X, axis=0)
S = X.T.dot(X)/(m-1)
print("Matriz de Covarianza:")
printArray(S)
print('_'*30)
#------------------------------
D, U = np.linalg.eig(S)
print('eigenvalores X.T X = ')
printArray(D,2)
print('eigenvectores X.T X= ')
printArray(U,2)
print('_'*30)
#------------------------------
U, D, V = np.linalg.svd(S, full_matrices=True)
print('SVD descomposición de X.T X : U D V')
printArray(U,2)
printArray(D,2)
printArray(V,2)
print('_'*30)#------------------------------
U, D, V = np.linalg.svd(X, full_matrices=True)
print('SVD descomposición de X ')
print('Eingenvectores de X X.T')
printArray(U,2)
print('Eingenvalores mayores que cero de (X X.T) o (X.T X)')
printArray(np.sqrt(D))
print('Eingenvectores de X.T X')
printArray(V, 2, False)
print('_'*30)
#-------------------------------------------------------------------
EigSvd(X)
Matriz de Covarianza:
[[ 1. -0.009 0.009]
[-0.009 1. 0.003]
[ 0.009 0.003 1. ]]
______________________________
eigenvalores X.T X =
[ 0.99 1.01 1. ]
eigenvectores X.T X=
[[-0.66 -0.75 -0.01]
[-0.52 0.45 0.73]
[ 0.54 -0.49 0.69]]
______________________________
SVD descomposición de X.T X : U D V
[[ 0.75 -0.01 -0.66]
[-0.45 0.73 -0.52]
[ 0.49 0.69 0.54]]
[ 1.01 1. 0.99]
[[ 0.75 -0.45 0.49]
[-0.01 0.73 0.69]
[-0.66 -0.52 0.54]]
______________________________
SVD descomposición de X
Eingenvectores de X X.T
[[ 0.02 -0. -0.02 ..., 0.01 0.02 -0. ]
[ 0.01 0. -0. ..., -0. 0.01 0.01]
[ 0.01 -0.02 0. ..., 0.01 0. 0.01]
...,
[-0.01 0.01 0. ..., 1. -0. -0. ]
[-0.02 -0.01 0.01 ..., -0. 1. -0. ]
[-0.01 -0. -0.01 ..., -0. -0. 1. ]]
Eingenvalores mayores que cero de (X X.T) o (X.T X)
[ 10.029 10.008 9.963]
Eingenvectores de X.T X
[[ 0.75 -0.45 0.49]
[ 0.01 -0.73 -0.69]
[-0.66 -0.52 0.54]]
______________________________
# Code source: Mariano Rivera
# mayo 2017
#-------------------------------------------------------------------
def pca_basico(X, varianzaExplicada=1):
X = X - np.mean(X, axis=0)
X = X / np.std(X, axis=0)
corrMat=np.cov(X, rowvar=False)
evals, evecs = np.linalg.eigh(corrMat)
print("_"*30)
print('PCA Código Básico')
print("_"*30)
print('Matriz de Correlación:')
printArray(corrMat)
evals, evecs = np.linalg.eigh(corrMat)
# ordenando los eigenvalores de mayor a menor
# np.linalg.eig no los devuelve ordenados
idx = np.argsort(evals)[::-1] # indices ordenados
evecsSorted = evecs[:,idx] # Eigenvalores ordenados
evalsSorted = evals[idx] # Eighenvectores ordenados
varianzaAcumulada = np.cumsum(evalsSorted)/np.sum(evalsSorted)
# indice de umbral de varianza retenida
indexVar=np.argmax(varianzaAcumulada>=varianzaExplicada)
evecsSorted = evecsSorted[:,:indexVar+1]
# datos proyectados y reducidos
Beta=np.dot(evecsSorted.T, X.T).T
print('Eigenvalores')
printArray(evalsSorted)
print('Eigenvectores')
printArray(evecsSorted)
print("_"*30)
#-------------------------------------------------------------------
def pca_with_sklean(X, varianzaExplicada=1.0):
print('PCA usando sklearn')
clf=PCA(varianzaExplicada)
X_train=X
X_train=clf.fit_transform(X_train)
print("_"*30)
print('Eigenvalores')
printArray(clf.explained_variance_)
print('Eigenvectores')
printArray(clf.components_)
print('Razón de explicación por componente:', clf.explained_variance_ratio_)
print(clf)
print("_"*30)
#-------------------------------------------------------------------
pca_basico(X, varianzaExplicada=.98)
pca_with_sklean(X, varianzaExplicada=0.98)
______________________________
PCA Codigo Básico
______________________________
Matriz de Correlación:
[[ 1. -0.009 0.009]
[-0.009 1. 0.003]
[ 0.009 0.003 1. ]]
Eigenvalores
[ 1.012 1.003 0.985]
Eigenvectores
[[ 0.749 -0.014 0.662]
[-0.447 0.727 0.521]
[ 0.489 0.687 -0.538]]
______________________________
PCA usando sklearn
______________________________
Eigenvalores
[ 1.012 1.003 0.985]
Eigenvectores
[[ 0.749 -0.447 0.489]
[ 0.014 -0.727 -0.687]
[ 0.662 0.521 -0.538]]
Razón de explicación por componente: [ 0.33718644 0.33441098 0.32840258]
PCA(copy=True, iterated_power='auto', n_components=0.98, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
______________________________
Distribución Gaussiana multivariada correlacionada
# Code source: Mariano Rivera
# mayo 2017
# Gaussiana tri-variada no-correlacionada
v= np.array([2,3,4])
v= v / np.sqrt(v.dot(v))
T = np.outer(v,v) +.2*np.eye(3) # tensor definido positivo simético
L, R = np.linalg.eig(T)
# R es la matriz de rotacion que nos interesa,
# L tienen eigenvalores delk tensor, pero vamos a reemplazarlos
# matriz de correlacion generada con varianzas
# (eigenvalores) igual al caso no correlacionado
CovC = R.dot(np.diag(D).dot(R.T))
print('Covarianza con datos correlacionados: Def. positiva y simétrica')
printArray(CovC)
D, U = np.linalg.eig(CovC)
print('Eigenvalores:')
printArray(D)
print('EigenVectores:')
printArray(U)
m = 300 # número de datos (tamaño de la mustra)
n, nn = CovC.shape # dimensión de cada dato
Med = np.array([1, 2, -10]) # vector de medias
X = np.random.multivariate_normal(Med, CovC, m)
Covarianza con datos correlacionados: Def. positiva y simétrica
[[ 1.325 -0.212 -0.299]
[-0.212 0.209 0.257]
[-0.299 0.257 0.367]]
Eigenvalores:
[ 1.471 0.41 0.019]
EigenVectores:
[[ 0.928 -0.371 0.007]
[-0.217 -0.557 -0.802]
[-0.301 -0.743 0.598]]
plotData3D(X)
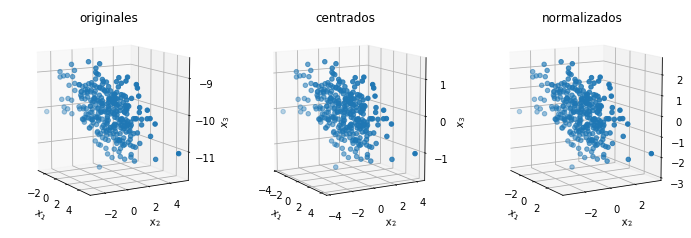
EigSvd(X)
Matriz de Covarianza:
[[ 1.003 -0.385 -0.397]
[-0.385 1.003 0.927]
[-0.397 0.927 1.003]]
______________________________
eigenvalores X.T X =
[ 2.19 0.75 0.08]
eigenvectores X.T X=
[[ 0.42 -0.91 0.01]
[-0.64 -0.31 -0.7 ]
[-0.64 -0.29 0.71]]
______________________________
SVD descomposición de X.T X : U D V
[[-0.42 -0.91 0.01]
[ 0.64 -0.31 -0.7 ]
[ 0.64 -0.29 0.71]]
[ 2.19 0.75 0.08]
[[-0.42 0.64 0.64]
[-0.91 -0.31 -0.29]
[ 0.01 -0.7 0.71]]
______________________________
SVD descomposición de X
Eingenvectores de X X.T
[[ 0. 0.01 0.02 ..., -0.01 0.03 0.01]
[-0.04 0.09 0.08 ..., 0.07 -0.06 0.17]
[-0.02 -0.04 -0.08 ..., -0.06 0.02 -0.08]
...,
[-0.06 -0.05 -0.05 ..., 0.99 0. -0.01]
[ 0.06 0.02 0.01 ..., 0.01 1. 0.01]
[-0.12 -0.13 -0.06 ..., -0.01 0.01 0.97]]
Eingenvalores mayores que cero de (X X.T) o (X.T X)
[ 5.057 3.864 2.187]
Eingenvectores de X.T X
[[-0.42 0.64 0.64]
[ 0.91 0.31 0.29]
[ 0.01 -0.7 0.71]]
______________________________
pca_basico(X, varianzaExplicada=.98)
pca_with_sklean(X, varianzaExplicada=0.98)
______________________________
PCA Código Básico
______________________________
Matriz de Correlación:
[[ 1.003 -0.385 -0.397]
[-0.385 1.003 0.927]
[-0.397 0.927 1.003]]
Eigenvalores
[ 2.188 0.746 0.077]
Eigenvectores
[[-0.423 0.906 0.011]
[ 0.64 0.307 -0.705]
[ 0.642 0.291 0.709]]
______________________________
PCA usando sklearn
______________________________
Eigenvalores
[ 2.181 0.743 0.076]
Eigenvectores
[[-0.423 0.64 0.642]
[-0.906 -0.307 -0.291]
[-0.011 0.705 -0.709]]
Razón de explicación por componente: [ 0.72688877 0.2476747 0.02543653]
PCA(copy=True, iterated_power='auto', n_components=0.98, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
______________________________
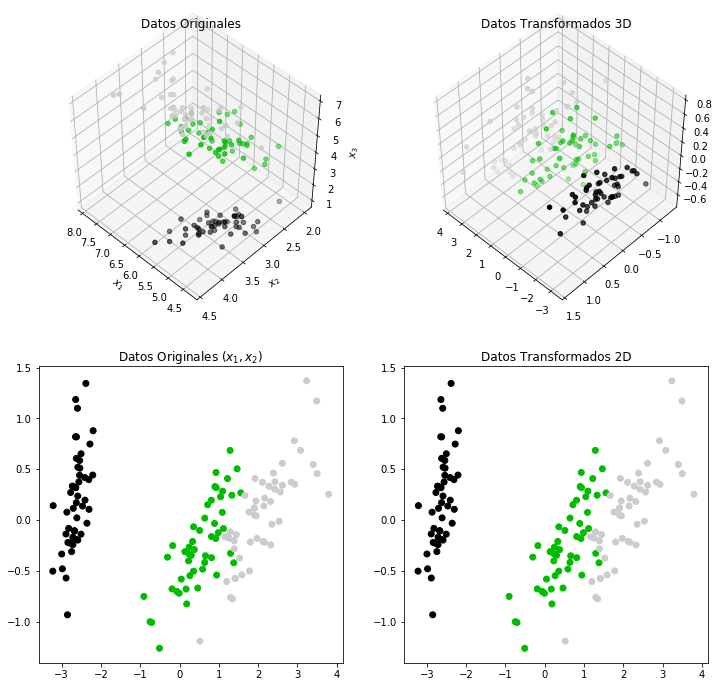
Ejemplo de reducción de Dimensionalidad usando PCA
Basado en el ejemplo de código mas elaborado en sklearn por Gaël Varoquaux, License BSD 3 clause.
# Code source: Mariano Rivera
# mayo 2017
import numpy as np
import matplotlib.pyplot as plt
from sklearn import decomposition
from sklearn import datasets
iris = datasets.load_iris()
X = iris.data
y = iris.target
pca = decomposition.PCA(n_components=3)
pca.fit(X)
Xt = pca.transform(X)
pca2 = decomposition.PCA(n_components=2)
pca2.fit(X)
Xt2 = pca2.transform(X)
fig = plt.figure(1, figsize=(12, 12))
ax = fig.add_subplot(221, projection='3d')
ax.scatter(X[:, 0], X[:, 1], X[:, 2], c=y, cmap=plt.cm.spectral)
ax.view_init(elev=48,azim=134)
ax.set_title('Datos Originales')
ax.set_xlabel('$x_1$')
ax.set_ylabel('$x_2$')
ax.set_zlabel('$x_3$')
ax = fig.add_subplot(222, projection='3d')
ax.scatter(Xt[:, 0], Xt[:, 1], Xt[:, 2], c=y, cmap=plt.cm.spectral)
ax.view_init(elev=48,azim=134)
ax.set_title('Datos Transformados 3D')
ax = fig.add_subplot(223) #, projection='2d')
ax.scatter(Xt[:, 0], Xt[:, 1], c=y, cmap=plt.cm.spectral)
#ax.view_init(elev=48,azim=134)
ax.set_title('Datos Originales $(x_1,x_2)$')
ax = fig.add_subplot(224) #, projection='2d')
ax.scatter(Xt2[:, 0], Xt2[:, 1], c=y, cmap=plt.cm.spectral)
#ax.view_init(elev=48,azim=134)
ax.set_title('Datos Transformados 2D')
plt.show()