Mínimo Cuadrados Generalizados
En el marco del modelo clásico, los supuestos de homocedasticidad, \(E\left(\epsilon_i^2\right)=\sigma^2\ \ (i=1,2, \ldots n)\), y ausencia de autocorrelación, \(E\left(\epsilon_i \epsilon_j\right)=0\) \(\ \forall i \neq j \ (i, j=1,2, \ldots n)\), implican que la matriz de varianzas y covarianzas de \(\boldsymbol{\epsilon}\) es
\[
\begin{align}
\operatorname{VarCov}(\boldsymbol{\epsilon}) & =\mathbb{E}\left[(\boldsymbol{\epsilon}-\mathbb{E}(\boldsymbol{\epsilon}))\left(\boldsymbol{\epsilon}-\mathbb{E}\left(\boldsymbol{\epsilon}\right)\right)^{\top}\right]=\mathbb{E}\left[\left(\begin{array}{c}
\epsilon_1 \\
\epsilon_2 \\
\vdots \\
\epsilon_n
\end{array}\right)\left(\begin{array}{llll}
\epsilon_1 & \epsilon_2 & \ldots & \epsilon_n
\end{array}\right)\right] \\
& =\left(\begin{array}{cccc}
\mathbb{E}\left(\epsilon_1^2\right) & \mathbb{E}\left(\epsilon_1 \epsilon_2\right) & \ldots & \mathbb{E}\left(\epsilon_1 \epsilon_n\right) \\
\mathbb{E}\left(\epsilon_2 \epsilon_1\right) & \mathbb{E}\left(\epsilon_2^2\right) & \ldots & \mathbb{E}\left(\epsilon_2 \epsilon_n\right) \\
\vdots & \vdots & \ddots & \vdots \\
\mathbb{E}\left(\epsilon_n \epsilon_1\right) & \mathbb{E}\left(\epsilon_n \epsilon_2\right) & \ldots & \mathbb{E}\left(\epsilon_n^2\right)
\end{array}\right)=\left(\begin{array}{cccc}
\sigma^2 & 0 & \ldots & 0 \\
0 & \sigma^2 & \ldots & 0 \\
\vdots & \vdots & \ddots & \vdots \\
0 & 0 & \ldots & \sigma^2
\end{array}\right)=\sigma^2 I_n
\end{align}
\]
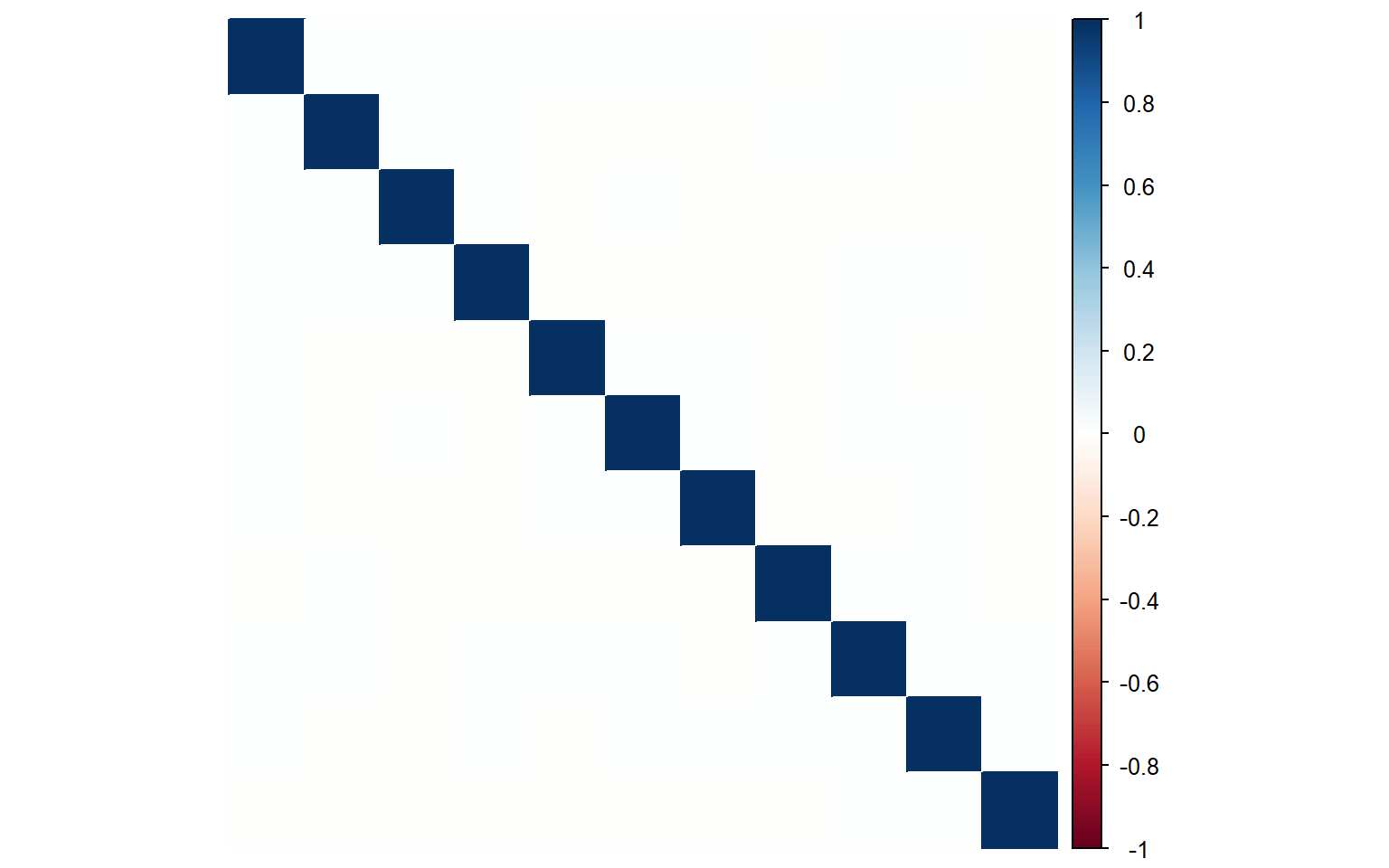
El supuesto de homocedasticidad implica que todos los elementos de la diagonal principal de \(\operatorname{VarCov}(\boldsymbol{\epsilon})\), las varianzas, son iguales a un escalar \(\sigma^2\), mientras que el de autocorrelación implica que los elementos situados fuera de la diagonal principal de \(\operatorname{VarCov}(\boldsymbol{\epsilon})\), las covarianzas, son iguales a cero. Si relajamos estos dos supuestos, entonces la matriz de varianzas y covarianzas deja de ser escalar
\[
\operatorname{VarCov}(\boldsymbol{u})=\left(\begin{array}{cccc}
\mathbb{E}\left(\epsilon_1^2\right) & \mathbb{E}\left(\epsilon_1 \epsilon_2\right) & \ldots & \mathbb{E}\left(\epsilon_1 \epsilon_n\right) \\
\mathbb{E}\left(\epsilon_2 \epsilon_1\right) & \mathbb{E}\left(\epsilon_2^2\right) & \ldots & \mathbb{E}\left(\epsilon_1 \epsilon_n\right) \\
\vdots & \vdots & \ddots & \vdots \\
\mathbb{E}\left(\epsilon_n \epsilon_1\right) & \mathbb{E}\left(\epsilon_n \epsilon_2\right) & \ldots & \mathbb{E}\left(\epsilon_n^2\right)
\end{array}\right)=\left(\begin{array}{cccc}
\sigma_1^2 & \sigma_{12} & \ldots & \sigma_{1 n} \\
\sigma_{21} & \sigma_2^2 & \ldots & \sigma_{2 n} \\
\vdots & \vdots & \ddots & \vdots \\
\sigma_{n 1} & \sigma_{n 2} & \ldots & \sigma_n^2
\end{array}\right)=\Sigma
\]
Definición 4.2 (El modelo lineal general con perturbaciones no esféricas) El modelo lineal general con perturbaciones no esféricas es \[
Y_i=\beta_1+\beta_2 X_{2 i}+\cdots+\beta_k X_{k i}+\epsilon_i, \quad i=1, \ldots, n
\] en donde \(\mathbb{E}\left(\epsilon_i\right)=0\), \(\mathbb{E}\left(\epsilon_i^2\right)=\sigma_i^2\) y \(\mathbb{E}\left(\epsilon_i \epsilon_j\right)=\sigma_{i j}\) \(\forall i \neq j\), que podemos escribir en notación matricial como \[
\boldsymbol{Y}=X \boldsymbol{\beta}+\boldsymbol{u},\quad \text{ con } \quad \mathbb{E}(\boldsymbol{\epsilon})=\boldsymbol{0}, \quad \mathbb{E}\left(\boldsymbol{\epsilon \epsilon}^{\top}\right) = \Sigma.
\]
Conviene escribir \(\Sigma=\sigma^2 \Omega\), para obtener el modelo lineal general con perturbaciones esféricas como un caso especial del modelo con perturbaciones no esféricas, \(\Omega = I_n\).
A continuación vamos a demostrar que en el modelo lineal general con perturbaciones no esféricas, hay un estimador alternativo y superior al estimador de mínimos cuadrados ordinarios: el estimador de mínimos cuadrados generalizados, que es equivalente al estimador de máxima verosimilitud cuando el vector de errores sigue una distribución normal multivariada con matriz de Var-Cov \(\Sigma\).
Estimador de mínimos cuadrados ordinarios (EMO)
Proposición 4.2 En el modelo lineal general con heterocedasticidad y/o autocorrelación, el estimador de mínimos cuadrados ordinarios es \[
\widehat{\boldsymbol{\beta}}_{\text{MCO}} = \left(X^{\top} X\right)^{-1} X^{\top} \boldsymbol{Y}.
\]
Esto es trivial demostrar ya que el método de mínimos cuadrados ordinarios no tiene en cuenta la matriz de varianzas y covarianzas de los errores al minimizar la suma de cuadrados de los residuos: \[
Q=(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}})^{\top}(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}}).
\]
Proposición 4.3 En el modelo lineal general con heterocedasticidad y/o autocorrelación, el estimador \(\text{MCO}\) es insesgado.
Esta proposición es lógica porque que la propiedad de insesgadez se basa en los supuestos de regresores no estocásticos y \(\mathbb{E}(\boldsymbol{\epsilon})=\boldsymbol{0}\), pero no tiene en cuenta la matriz de varianzas y covarianzas de los errores. Así que \[
\mathbb{E}\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}} \right) =\left[\left(X^{\top} X\right)^{-1} X^{\top} (X\boldsymbol{\beta})\right]=\boldsymbol{\beta}.
\]
Proposición 4.4 En el modelo lineal general con heterocedasticidad y/o autocorrelación, la matriz de varianzas y covarianzas de \(\widehat{\boldsymbol{\beta}}_{\text{MCO}}\) es \[
\operatorname{Var}\left(\widehat{\boldsymbol{\beta}}_{M C O}\right)=\sigma^2\left(X^{\top} X\right)^{-1}\left(X^{\top} \Omega X\right)\left(X^{\top} X\right)^{-1}
\]
Por definición \[
\operatorname{VarCov}\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}\right)=\mathbb{E}\left[\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}-\mathbb{E}\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}\right)\right)\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}-\mathbb{E}\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}\right)\right)^{\top}\right].
\]
Como el estimador es insesgado, \[
\begin{align}
\widehat{\boldsymbol{\beta}}_{\text{MCO}}-\mathbb{E}\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}\right)=\widehat{\boldsymbol{\beta}}_{\text{MCO}}-\boldsymbol{\beta} &= \left(X^{\top} X\right)^{-1} X^{\top}\boldsymbol{Y}-\boldsymbol{\beta}\\
&=\left(X^{\top} X\right)^{-1} X^{\top}(\boldsymbol{Y}-X\boldsymbol{\beta})\\
&=\left(X^{\top} X\right)^{-1} X^{\top} \boldsymbol{\epsilon}.
\end{align}
\] Entonces \[
\begin{aligned}
\operatorname{VarCov}\left(\widehat{\boldsymbol{\beta}}_{\text{MCO}}\right) & =\mathbb{E}\left[\left(X^{\top} X\right)^{-1} X^{\top} \boldsymbol{\epsilon} \boldsymbol{\epsilon}^{\top} X\left(X^{\top} X\right)^{-1}\right]=\left(X^{\top} X\right)^{-1} X^{\top} \mathbb{E}\left[\boldsymbol{\epsilon} \boldsymbol{\epsilon}^{\top}\right] X\left(X^{\top} X\right)^{-1} \\
& =\left(X^{\top} X\right)^{-1} X^{\top}\left[\sigma^2\ \Omega\right] X\left(X^{\top} X\right)^{-1}
=\sigma^2\left(X^{\top} X\right)^{-1}\left(X^{\top} \Omega X\right)\left(X^{\top} X\right)^{-1}
\end{aligned}
\]
Proposición 4.5 (Distribución del estimador) Si suponemos que \(\boldsymbol{\epsilon} \sim N\left(\mathbf{0}, \sigma\ \Omega\right)\), el estimador MCO tiene una distribución normal \[
\widehat{\boldsymbol{\beta}}_{\text{MCO}} \sim N\left(\boldsymbol{\beta},\ \ \sigma^2 \ \left(X^{\top} X\right)^{-1}\left(X^{\top} \Omega X\right)\left(X^{\top} X\right)^{-1}\right).
\]
Proposición 4.6 El estimador \(\hat{\sigma}^2=\boldsymbol{e}^{\top} \boldsymbol{e} /(n-p)\) es un estimador sesgado.
\[
\begin{align}
\mathbb{E}\left(\boldsymbol{e}^{\top} \mathbf{e}\right)&=\mathbb{E}\left(\boldsymbol{Y}^{\top} (I_n-P) \boldsymbol{Y}\right)\\
&=\operatorname{tr}\left[(I_n-P)\Sigma\right]+(X\boldsymbol{\beta})^{\top}(I_n-P) (X\boldsymbol{\beta}),\quad \text{donde }\ \Sigma=\sigma^2\Omega\\
&=\sigma^2\operatorname{tr}\left[(I_n-P)\Omega\right]+0\\
&\neq \sigma^2(n-p), \qquad \text{ en general.}
\end{align}
\]
El estimador de mínimos cuadrados generalizados
Ahora planteamos la siguiente pregunta: ¿es posible transformar un modelo lineal general con perturbaciones no esféricas en un modelo lineal general con perturbaciones esféricas?
Si la respuesta es afirmativa, entonces el modelo transformado cumplirá las hipótesis básicas y todos los resultados establecidos en los temas anteriores serán de aplicación directa. El estimador de mínimos cuadrados ordinarios (MCO) en el modelo transformado se denomina estimador de mínimos cuadrados generalizados (MCG).
Para encontrar un modelo transformado con las hipótesis básicas, premultiplicamos el modelo lineal general con perturbaciones no esféricas por una matriz \(T\) no estocástica
\[
T\boldsymbol{Y}=TX \boldsymbol{\beta}+T\boldsymbol{\epsilon}
\]
Este modelo transformado puede escribirse como \[
\boldsymbol{Y}_*=X_* \boldsymbol{\beta}+\boldsymbol{\epsilon}_*
\]
El término de error en el modelo transformado \(\boldsymbol{\epsilon}_*\) cumple las siguientes propiedades:
- \(\mathbb{E}\left(\boldsymbol{\epsilon}_*\right)=\mathbb{E}(T\boldsymbol{\epsilon})=T \mathbb{E}(\boldsymbol{\epsilon})=\boldsymbol{0}\)
-
\(\mathbb{E}\left(\boldsymbol{\epsilon}_* \boldsymbol{\epsilon}_*^{\top}\right)=\mathbb{E}\left(T\boldsymbol{\epsilon \epsilon}^{\top} T^{\top}\right)=T \mathbb{E}\left(\boldsymbol{\epsilon \epsilon}^{\top}\right) T^{\top} = \sigma^2\ T \Omega T^{\top}\).
Si la matriz \(T\) es tal que \(\sigma^2\, T\, \Omega\, T^{\top} = \sigma^2 I_n\), entonces el modelo transformado:
- contiene los parámetros de interés \(\boldsymbol{\beta}\) y \(\sigma\)
- cumple las hipótesis básicas.
De aquí, el estimador de mínimos cuadrados ordinarios en el modelo transformado proporciona el estimador lineal, insesgado y eficiente de \(\boldsymbol{\beta}\) y el estimador insesgado de \(\sigma^2\).
Proposición 4.7 (Existencia) Existe una matriz \(T\) tal que \(T\,\Omega\, T^{\top} = I_n\)
Usando el teorema espectral, podemos escribir \[
\Omega \, C = C \, \Lambda
\] en donde \(\Lambda=\operatorname{diag}\left(\lambda_1, \ldots, \lambda_n\right)\) es la matriz diagonal de eigenvalores y \(C\) es la matriz ortogonal con los eigenvectores. De aquí, podemos escribir \[
\Omega = C \Lambda C^{\top}
\]
Como \(\Lambda^{1/2}=\operatorname{diag}\left(\sqrt{\lambda_1}, \ldots, \sqrt{\lambda_n}\right)\), tenemos que
\[
\Omega = C \Lambda^{1 / 2} \Lambda^{1 / 2} C^{\top}
\]
Premultiplicando \(\Omega\) por \(\Lambda^{-1 / 2} C^{\top}\) y postmultiplicando por \(C \Lambda^{-1 / 2}\), \[
\Lambda^{-1 / 2} C^{\top} \Omega C \Lambda^{-1 / 2} = \Lambda^{-1 / 2} C^{\top} C \Lambda^{1 / 2} \Lambda^{1 / 2} C^{\top} C \Lambda^{-1 / 2}
= I_n
\] De aquí, vemos que la matriz buscada es \[
T = \Lambda^{-1 / 2} C^{\top}.
\]
De la demostración anterior, se derivan las dos siguientes relaciones que son de interés:
-
\(\Omega^{-1}= T^{\top} T\) \[
\begin{align}
\because \ \Omega^{-1}&=C\Lambda^{-1}C^{\top}\\
&=C\Lambda^{-1/2}\Lambda^{-1/2}C^{\top}\\
&=(\Lambda^{-1/2}C^{\top})^{\top}\Lambda^{-1/2}C^{\top}
\end{align}
\]
-
\(\Omega = T^{-1} \left(T^{\top}\right)^{-1}\) \[
\begin{align}
\because \ T^{-1} \left(T^{\top}\right)^{-1}&=C^{\top}\Lambda^{1/2}\left(C\Lambda^{-1/2}\right)^{-1}\\
&=C^{\top}\Lambda^{1/2}(\Lambda^{1/2})C^{\top},\quad \text{porque }\ C^{-1}=C^{\top}.
\end{align}
\]
Proposición 4.8 (estimador de Mínimos Cuadrados Generalizados, MCG) El estimador lineal, insesgado y óptimo de \(\boldsymbol{\beta}\) es \[
\widehat{\boldsymbol{\beta}}_{\text{MCG}}=\left(X^{\top} \boldsymbol{\Omega}^{-1} X\right)^{-1} X^{\top} \Omega^{-1} \mathbf{Y}
\] que se denomina estimador de Mínimos Cuadrados Generalizados o estimador de Aitken.
Como el modelo transformado cumple los supuestos del modelo clásico, el estimador el estimador de mínimos cuadrados ordinarios \[
\widehat{\boldsymbol{\beta}}=\left(X_*^{\top} X_*\right)^{-1} X_*^{\top} \mathbf{Y}_*
\] será el estimador lineal, insesgado y óptimo, que podemos expresarse en términos de los datos originales \[
\widehat{\boldsymbol{\beta}}=\left(X^{\top} T^{\top} T X\right)^{-1} X^{\top} T^{\top} T \, \mathbf{Y}=\left(X^{\top} \Omega^{-1} X\right)^{-1} X^{\top} \Omega^{-1} \mathbf{Y}.
\]
Proposición 4.9 La matriz de varianzas y covarianzas del estimador de MCG es
\[
\operatorname{Var}\left(\widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)=\sigma^2\left(X^{\top} \Omega^{-1} X\right)^{-1}
\]
La matriz de varianzas y covarianzas del estimador de MCO de \(\boldsymbol{\beta}\) en el modelo transformado es \[
\operatorname{VarCov}\left(\widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)=\sigma^2\left(X_*^{\top} X_*\right)^{-1}=\sigma^2\left(X^{\top} T^{\top} T X\right)^{-1}=\sigma^2\left(X^{\top} \Omega^{-1} X\right)^{-1}.
\]
El Teorema de Gauss-Markov aplicado en el modelo transformado, garantiza que el estimador MCO es el estimador con varianza m¶‡nima entre los estimadores lineales e insesgados.
Proposición 4.10 El estimador insesgado de \(\sigma^2\) es \[
\widehat{\sigma}_{\text{MCG}}^2=\frac{\left(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)^{\top} \Omega^{-1}\left(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)}{n-p}
\]
El estimador insesgado de \(\sigma^2\) en el modelo transformado es \[
\begin{align}
\widehat{\sigma}^2 &=\frac{\left(\boldsymbol{Y}_*-X_* \widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)^{\top}\left(\boldsymbol{Y}_*-X_* \widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)}{n-p}
=\frac{\left(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)^{\top} T^{\top}T\left(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}}_{\text{MCG}}\right)}{n-p}.
\end{align}
\]
Estimadores máximo verosímiles
Como \(\boldsymbol{Y}\sim N_n(X\boldsymbol{\beta}, \sigma^2\Omega)\), entonces \[
\ell\left(\boldsymbol{\beta}, \sigma^2\right)=-\frac{n}{2} \ln (2 \pi)-\frac{n}{2} \ln \left(\sigma^2\right)-\frac{1}{2} \ln (\Omega)-\frac{1}{2 \sigma^2}(\boldsymbol{Y}-X \boldsymbol{\beta})^{\top} \Omega^{-1}(\boldsymbol{Y}-X \boldsymbol{\beta}),
\] ya que \(\left|\,\sigma^2 \Omega\,\right|=\left(\sigma^2\right)^n|\Omega|\).
Proposición 4.11 (EMVs) Los estimadores de máximo verosímiles de \(\boldsymbol{\beta}\) y \(\sigma_u^2\) son \[
\begin{aligned}
\widehat{\boldsymbol{\beta}} & =\left(X^{\top} \Omega^{-1} X\right)^{-1} X^{\top} \Omega^{-1} \boldsymbol{Y} \\[5pt]
\widehat{\sigma}^2 & =\frac{(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}})^{\top} \Omega^{-1}(\boldsymbol{Y}-X \widehat{\boldsymbol{\beta}})}{n}
\end{aligned}
\]
Bajo el supuesto normalidad, es estimador de mínimos cuadrados generalizados coincide con el estimador de máxima verosimilitud.
La demostración es trivial a partir de obtener las derivadas parciales de \(\ell\left(\boldsymbol{\beta}, \sigma^2\right)\) respecto a \(\boldsymbol{\beta}\) y \(\sigma^2\): \[
\begin{aligned}
& \frac{\partial \ell\left(\boldsymbol{\beta}, \sigma^2\right)}{\partial \boldsymbol{\beta}}=-\frac{1}{\sigma^2}\left(-X^{\top} \Omega^{-1} \boldsymbol{Y}+X^{\top} \Omega^{-\mathbf{1}} X \boldsymbol{\beta}\right) \\
& \frac{\partial \ell\left(\boldsymbol{\beta}, \sigma^2\right)}{\partial \sigma^2}=-\frac{n}{2 \sigma^2}+\frac{1}{2 \sigma^4}(\boldsymbol{Y}-X \boldsymbol{\beta})^{\top}\Omega^{-1}(\boldsymbol{Y}-X \boldsymbol{\beta})
\end{aligned}
\]
igualandolas a cero y resoviendo simultáneamente.
El estimador de \(\text{MCG}\) supone que la matriz \(\Omega\) es conocida.
Mínimo Cuadrados Generalizados Factibles
Hasta ahora hemos supuesto que conocemos \(\Sigma=\sigma^2 \Omega\) o al menos \(\Omega\). El estimador de MCG en este caso es lineal, insesgado y de varianza mínima. Pero en la práctica la mayoría de las veces \(\Omega\) o \(\Sigma\) son desconocidas. En este caso el estimador MCG no es directamente calculable. La solución habitual es sustituir \(\Omega\) (o \(\Sigma\) ) por una estimación suya en la expresión del estimador de MCG dando lugar al estimador MCGF: \[
\begin{aligned}
\widehat{\boldsymbol{\beta}}_{\text{MCGF}} & =\left(X^{\top} \widehat{\Omega}^{-1} X\right)^{-1} X^{\top} \widehat{\Omega}^{-1} \boldsymbol{Y} \\
& =\left(X^{\top} \widehat{\Sigma}^{-1} X\right)^{-1} X^{\top} \widehat{\Sigma}^{-1} \boldsymbol{Y}.
\end{aligned}
\]
Bibliografía:
- Christensen, R. (2011). Plane answers to complex questions: The Theory of Linear Models. 4th Ed. New York: Springer.
- Seber, G. A., & Lee, A. J. (2003). Linear regression analysis. John Wiley & Sons.
- Radhakrishna R., Toutenburg, H.(1997) Linear Models Least Squares and Alternatives. Springer. (Cap 3.10)
Referencias:
- Box, G.E.P. & Cox, D.R. (1964) Analysis of Transformations. JRSS-B, Vol.26, No.2, 211-252.
- Thome, N. (2019). La inversa generalizada de Moore-Penrose y Aplicaciones. Publicaciones electrónicas Sociedad Matemática Mexicana, 21, 81. Liga a ebook
- Bott, R. y Duffin, R. J. (1953)On the algebra of networks, Trans. Amer. Math. Soc. 74:99-109.
- Drazin, M. P. (1958). “Pseudo-inverses in associative rings and semigroups”. The American Mathematical Monthly. 65 (7): 506–514.
- Rawlings, J. O., Pantula, S. G., & Dickey, D. A. (Eds.). (1998). Applied regression analysis: a research tool. New York, NY: Springer New York.
- Jolliffe, I. T. (1972). Discarding variables in a principal component analysis. I: Artificial data. Journal of the Royal Statistical Society Series C: Applied Statistics, 21(2), 160-173.